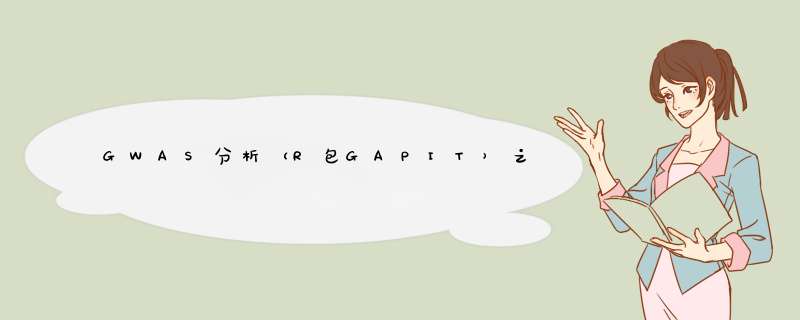
可以接受 standard HapMap format 或 numeric format。
第一种,Hapmap Format。
在基因型数据前有11列数据。尽管这11列数据都必须有,但读取的时候只读取第1、3、4列数据,其余数据可填充为NA。另,基因型数据可以是两个碱基形式的,也可以是单碱基形式的,如下表所示:
第二种,Numeric format。
这种格式的需要两个文件,一个指定SNP名称、染色体、和位置;一个指定所有个体在该位点的基因型。
无表头数据,表示亲缘关系的一个矩阵。
一个包含协变量的文件,包括群体结构之类的信息。第一列是个体名称,后面每一列都是协变量的值。
具体 *** 作:
表型文件不多赘述,笔者重复一遍 numeric format 格式的基因型文件的生成方式。
上述 *** 作会生成 3 个文件,分别是 xx_matrix.012、xx_matrix.012.pos、xx_matrix.012.indv。
SNP_INFORMATION的文件处理完成,接下来处理基因型文件,vcftools 直接生成的文件如下图所示:每一行表示一个个体,每一列表示一个位点,第一列是个体名称。
接下来,首先替换个体名称,利用 xx_matrix.012.indv 文件。
然后给每一列变量增加名称:我的 *** 作是先生成变量名称的文件。
以上,生成计算所需基因型文件。
相关链接:
GWAS分析(R包GAPIT)之一(相关包的安装)
GWAS分析(R包GAPIT)之二(数据准备)
GWAS分析(R包GAPIT)之三(计算运行)
原理如下图:
常用的GWAS芯片大约60万个位点,经过质控后大约只剩下30多万个位点,对于全基因组30亿个碱基来说,只覆盖了全基因组万分之一的区域,因此大片的区域为空白。经过基因型填补后,SNP密度大大增加,如果与表型相关联的位置没有SNP,填补前是没有显著性的,填补后则有可能出现显著性。因此,基因型填补可以大大增加GWAS的统计效能。
由于以上步骤很多,为了方便imputation过程,很多人开发了自动化的pipeline,如
待完善
参考:
http://www.bbmriwiki.nl/wiki/Impute2Pipeline
http://databeauty.com/blog/tutorial/2017/02/20/GWAS-prephasing-and-imputation.html
欢迎分享,转载请注明来源:内存溢出

 微信扫一扫
微信扫一扫
 支付宝扫一扫
支付宝扫一扫
评论列表(0条)