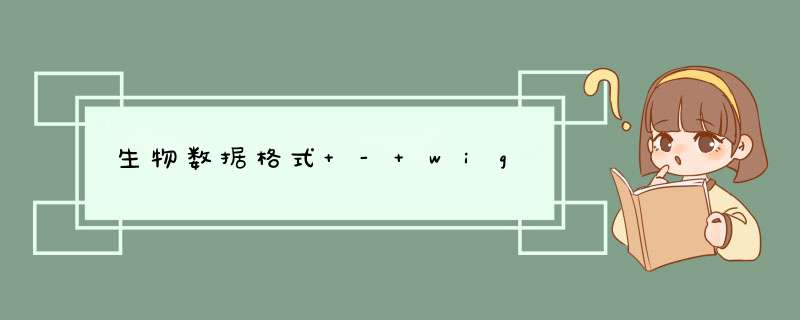
Wig的数据包括track line和data line,track line 定义了track的属性,比如track type=wiggle_0,指定track为Wig track。data line 主要由两部分格式组成,variableStep format和fixedStep format。variableStep format以一个声明开始,明确了染色体的序号,跨度(span)。后面跟两列数据,染色体开始的碱基位置,数据的值value(可以理解为覆盖度)。span参数可以将含有相同value的连续碱基包含在一起,使数据更加简洁。第二部分为fixedStep format, 由声明和单列数据组成。声明部分和variableStep format中各变量的意义一样。wig中的value值可以是整数,实数,正数或者负数。只有指定的位置有value值,没有制定的位置则没有value,且不会在UCSU Genome Browser中作出图。
关于wig格式文件的解读:如图,variableStep format span=150,包含的第一行数据“49304701 10.0”表示49304701-49304851有相同清镇手的value,为10.0,随后同理;第二部分fixeStep format表示第19号染色体493077401-493077600,493077701-493077900,493078001-493078200三个区域的value值分别为1000、900和800。
BigWig格式是wig格式文件的二进制压缩版本,用于密集连续的数据,并在基因组浏览器中进行可视化,是UCSC推荐的一种格式。但是wig格式要求数据必须是连续的并且由大小相等的元素组成,如果数据是稀疏或包含大小不同的元素时,则可以采用edGraph格式储存数据。BedGraph 的trace type和Wig文件很像,不过后面的数据和答嫌bed文件很类似,包含染色体号、起始位置、终止位置以及value。
USUC提供跨平台的 系列工具 对这三种文件格式进行相互转换与处理。常用工具包括:WigTobigWig、bigWigToWig、bigWigToBedGraph、bigWigSummary、bigWigAverageOverBed、bigWigInfo等。旅搭
前言
我们比对完fastq文件后,我们可以拿到bam文件,但是bam文件非常的大,处理起来很不方便。如果我们只是想知道这些read都比对到了基因组的什么区域,以及基因组上每个区域有多少read存在,此时用wig/bw/bdg则会更加方便。
如果没有这些工具,其实对我们的bam文件,用samtools软件也可以很容易得到基因组区域的覆盖度和测序深度,比如:
这其实就是wig文件的雏形,但是wig文件会更复杂一点!
它不需要第一列了,因为全部是重复字段,只需要在每个染色体的第一行定义好染色体即可。下面我们来说明这些不同的格式是怎么记录测序深度的。
wig文件全称叫Wiggle Track Format,用来绘制基因组上的图形轨迹的文件格式。wig格式是较老的格式,用来显示密集且连续的数据,比如GC含量,概率分数,转录组数据等。
wig数据有两种类型:
variableStep:基因组window大小不定
fixedStep:基山哪乎因组window大小相同
声明行:
单词variableStep开头
染色体
window大小(span):基因组上window大小,默认为1
数据行:两列,分别包含染色体和测序深度值。
举例说明:
1.2 fixedStep格式wig
声明行:
单词fixedStep开头
染色体
起始坐标
步长(step):相邻window起点之间的距离
window大小(span):基因组上window大小,默认为1
数据行:一列,包含测序深度值。
举例说明:
上述介绍了wig数据部分,其实wig还有一部分是设置展示图形的参数,这里不做过多解释。
UCSC提供了一个wig文件: http://genome.ucsc.edu/goldenPath/help/examples/wiggleExample.txt
bigwig格式文件就没什么好讲的了,它就是wig格式文件的二进制压缩版本,这样更加节省空间。
我们只需要用UCSC提供的工具把自己的wig文件转换一下即可。
wigToBigWig: http://hgdownload.cse.ucsc.edu/admin/exe/linux.x86_64/wigToBigWig
BedGraph格式文件,它是BED文件的扩展,是4列的BED格式(正常BED文件只有3列或者6列),但是需要添加UCSC的Genome Browser工具里面显示的属性,但是一般就定义有限的几个属性缓陆即可。
3.1 属性设置
和bed文件基本一致,只是多了:
最后第1行的设置信息
第4列的测序深逗悉度信息
4.1 bam文件转化为bigwig
将bam文件转为bw文件,有很多工具可以实现这个目标,但是我们这里只记录deeptools:
4.2 wig/bw/bedgraph相互转换工具
bedGraphToBigWig :Convert a bedGraph file to bigWig format. http://hgdownload.cse.ucsc.edu/admin/exe/linux.x86_64/bedGraphToBigWig
bigWigToBedGraph :Convert from bigWig to bedGraph format. http://hgdownload.cse.ucsc.edu/admin/exe/linux.x86_64/bigWigToBedGraph
bigWigToWig :Convert bigWig to wig. http://hgdownload.cse.ucsc.edu/admin/exe/linux.x86_64/bigWigToWig
bigWigSummary :Extract summary information from a bigWig file. http://hgdownload.cse.ucsc.edu/admin/exe/linux.x86_64/bigWigSummary
bigWigAverageOverBed :Compute average score of big wig over each bed, which may have introns. http://hgdownload.cse.ucsc.edu/admin/exe/linux.x86_64/bigWigAverageOverBed
bigWigInfo :Print out information about bigWig file. http://hgdownload.cse.ucsc.edu/admin/exe/linux.x86_64/bigWigInfo
分两步进行:将bigwig文件转为bdg文件,再将bdg文件转为bed文件
欢迎分享,转载请注明来源:内存溢出

 微信扫一扫
微信扫一扫
 支付宝扫一扫
支付宝扫一扫
评论列表(0条)