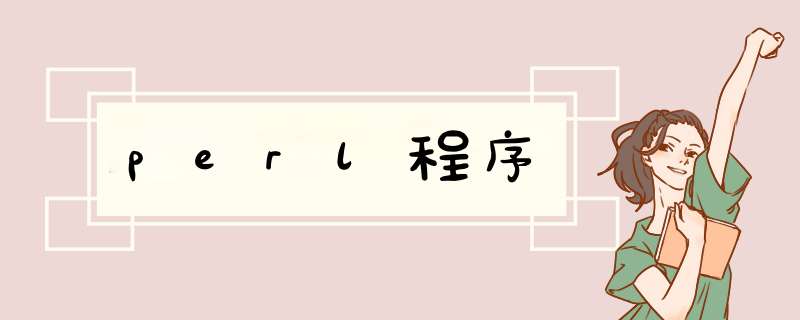
use strict
use warnings
my %flag_num=('myb'=>1,'erf'=>2,'abf'=>3,'dre'=>4,'myc'=>5,'bzip'=>6)
my $flag_count=6
my %stat
while(<>){
my ($name,$flag) = split
my $num=$flag_num{$flag}
$stat{$name}{$num}=1
}
my $subiter_array=make_subiter($flag_count)
my @data
for my $r (@$subiter_array){
my ($k, $keys_ref) = @$r
push @data,[$k,stat_step(\%stat,$keys_ref)]
}
my @sort_data=sort {
length($a->[0]) <=>length($b->[0])
or $a->[0] cmp $b->[0]
} @data
print join(" ",@$_),"\n" for @sort_data
sub make_subiter {
my ($n) = @_
my @keys_iter
my $num=2**$n-1
for my $i (1..$num){
my $j= sprintf("%b",$i)
my @temp=reverse split("",$j)
my @temp_key=map {$_+1} grep { $temp[$_] >0 } (0..$#temp)
my $key =join(":",@temp_key)
push @keys_iter,[$key,\@temp_key]
}
return \@keys_iter
}
sub stat_step {
my ($stat_ref,$keys_ref)=@_
my $keys_num=scalar(@$keys_ref)
my $count=0
while(my ($name,$r)=each %$stat_ref){
my $match = grep { exists $r->{$_} } @$keys_ref
$count++ if($match == $keys_num)
}
return $count
}
Perl优势是字符串处理。曾经是CGI的默认语言,但他的缺点是相对他的应用来说过于强大……在高速的发展中写CGI显得杀鸡用牛刀,然后要更快的有PHP,要更安全的用可编译的写CGI……所以似乎写CGI的不多了,除了我这些偏执狂……不过Perl在有些方面还是保持着生命力。例如,他是Linux默认的编译器之一,还是有软件例如VMWare Linux版是Perl的;在一些系统管理上可以当做强大的脚本工具……至少比你写C来得方便。还有就是最牛B的应用:Perl的模块,例如BioPerl是生物信息学的必备工具,其地位如同MatLab在数学中的地位……
这是对一个内存有行星名称星座亮度等内容的文件进行编辑的程序。第一段是用来查询一些名称对应亮度的。第二段是统计同一星座星数的,可以比较容易改成调用子程序的。第三段是对文件编辑后存入文件。不知道合不合要求
#!/usr/bin/perl -w
use strict
open F, "star25.txt" or die "Can't open file"
my @lines = <F>
close F
chomp @lines
s/#.*// foreach @lines
my (%sname, %spectrum, @fields)
foreach (grep !/^$/, @lines) {
@fields = split /:/, $_
next if @fields <6
$sname{$fields[0]} = $fields[1]
$spectrum{$fields[0]} = $fields[5]
}
print exists $sname{$_} ?
"$_: Scientific name is $sname{$_}, Spectrum is $spectrum{$_}\n" :
"$_: Not find!\n"
foreach ('Pole star', 'Castor', 'Altair', 'Hadar')
###################
my $c
foreach $c ('Ori', 'Cen', 'Cru') {
print "Number of starts in $c is ",
scalar(grep {$_ eq $c} map ((split / /, $_)[1], values %sname)),
"\n"
}
###################
my %Nspec
$Nspec{substr $_, 0, 1}++ foreach values %spectrum
open F, ">OBAFGKM.txt" or die "Can't open file for write."
print F "$_:$Nspec{$_}\n" foreach keys %Nspec
close F
1
欢迎分享,转载请注明来源:内存溢出

 微信扫一扫
微信扫一扫
 支付宝扫一扫
支付宝扫一扫
评论列表(0条)