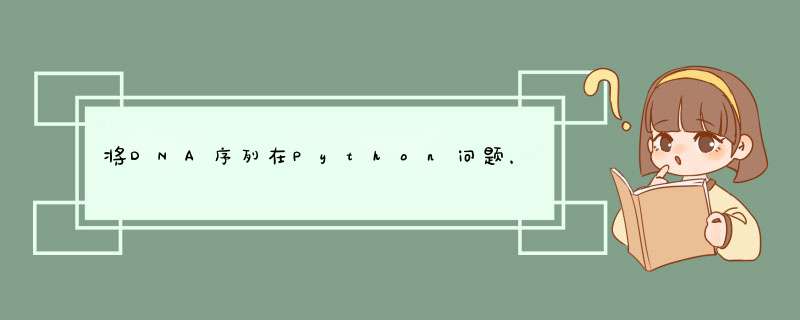
2 #-*- coding:utf-8 -*-
3 "将DNA序列转换为RNA序列,即将T转换为U即可,利用字符串的replace方法"
5 f=open('./test.txt', 'r')
6 line=f.read()
7 dna2rnaline=line.replace('T', 'U')
8 f.close()
9 f=open('./test.txt', 'w')
10 line=f.write(dna2rnaline)
11 f.close()#了解DNA序列和RNA序列的碱基差别
在处理基因组信息的时候,我们时不时需要对DNA序列进行处理,比如将DNA转换成RNA,对于较少批量的数据来说,可以人工处理,但是在面对大的fastq文件的时候,会显得苍白无力,下面分别介绍两种处理方式
1.单个序列
2.文件处理
以上小程序可以进行转换。
破译的过程其实挺简单 现在我们知道,DNA的信息储存是由3连密码子储存的,总共四种核苷酸,在DNA里是A T C G 在RNA里是A U C G 在转录的时候T和U是对等的,所以我们可以把它也看成是一种核苷酸.它们每三个一组,通过不同的排列组合方式,表达一种氨基酸,所以基因链可以通过读取三连密码子,来进行破译.在最初破译三连密码子的时候,有一个确定的方向,就是肯定一定数量的核苷酸的排列组合,对应的一个氨基酸信息,方向确定之后,接下来的工作就是确定密码子的数量,也就是说,几个碱基对应一个氨基酸,现已知道构成蛋白质的氨基酸共20种,那么四种碱基不可能一一对应,如果是2种碱基排列,则有16种组合,也不够,那么接下来就是3种碱基的排列,总共有64种组合,可以完全覆盖20种氨基酸,如果是4种碱基,则有256种组合,虽然也完全覆盖了20种氨基酸,但是数量太过悬殊,从一切节约的生命原则来看,未免信息量过大,会造成信息储存的传递的负担.所以当初的科学家暂定是3种碱基的组合为一个密码子.说实话,这有些运气的成分.当然,这种运气是被后来的事实验证了的.接下来就是确定各种碱基组合分别对应的是哪种氨基酸,这是个繁琐的工作,其实原理很简单,就是人工合成一段DNA,然后用来表达,看这段DNA序列最后合成的是哪种氨基酸.比如 首先要确定的是密码子“AAA”的信息 那么我们就合成一段序列“AAA AAA AAA AAA AAA AAA AAA”将其翻译成蛋白之后,发现这一段序列由7个赖氨酸组成,那么就可以相信,赖氨酸是由三个A编码的.当然,用64个密码子表示20种氨基酸,肯定会有重复,这就是密码子的简并性,就是会有多个密码子表示一个氨基酸,具体就不细说了.欢迎分享,转载请注明来源:内存溢出

 微信扫一扫
微信扫一扫
 支付宝扫一扫
支付宝扫一扫
评论列表(0条)