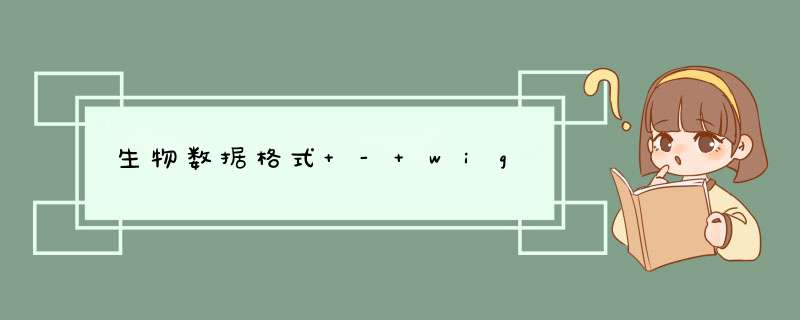
Wig的数据包括track line和data line,track line 定义了track的属性,比如track type=wiggle_0,指定track为Wig track。data line 主要由两部分格式组成,variableStep format和fixedStep format。variableStep format以一个声明开始,明确了染色体的序号,跨度(span)。后面跟两列数据,染色体开始的碱基位置,数据的值value(可以理解为覆盖度)。span参数可以将含有相同value的连续碱基包含在一起,使数据更加简洁。第二部分为fixedStep format, 由声明和单列数据组成。声明部分和variableStep format中各变量的意义一样。wig中的value值可以是整数,实数,正数或者负数。只有指定的位置有value值,没有制定的位置则没有value,且不会在UCSU Genome Browser中作出图。
关于wig格式文件的解读:如图,variableStep format span=150,包含的第一行数据“49304701 100”表示49304701-49304851有相同的value,为100,随后同理;第二部分fixeStep format表示第19号染色体493077401-493077600,493077701-493077900,493078001-493078200三个区域的value值分别为1000、900和800。
BigWig格式是wig格式文件的二进制压缩版本,用于密集连续的数据,并在基因组浏览器中进行可视化,是UCSC推荐的一种格式。但是wig格式要求数据必须是连续的并且由大小相等的元素组成,如果数据是稀疏或包含大小不同的元素时,则可以采用edGraph格式储存数据。BedGraph 的trace type和Wig文件很像,不过后面的数据和bed文件很类似,包含染色体号、起始位置、终止位置以及value。
USUC提供跨平台的 系列工具 对这三种文件格式进行相互转换与处理。常用工具包括:WigTobigWig、bigWigToWig、bigWigToBedGraph、bigWigSummary、bigWigAverageOverBed、bigWigInfo等。最近做的项目要对 reference genome 基于突变进行一些modify,制作 personalized genome 或者说是 psuedo-genome 伪基因组。其实就是把某个测序样本call出来的 SNP && indel 替换掉参考基因组对应位置的碱基。
自己可以编写脚本修改,使用 Perl 中的 substr 来进行单个位点修改,把坐标按照从后往前的顺序,而不是从前到后。
其实有蛮多工具可以做这个的,在这里安利一下,大家可以使用体验一下。
GATK作为通用的变异检测的软件,其中有很多有用的工具,这里介绍一下 FastaAlternateReferenceMaker : Create an alternative reference by combining a fasta with a vcf
准备好检测的 VCF 文件,参考基因组FASTA文件,使用如下命令:
这里的 -L 参数可以多单个基因或者基因组一段区域进行替换。
--snp-mask 参数,当构建时 psuedo-genomefa 时,该VCF文件中的SNP用作掩码(在序列中插入N)。
运行结束,个人的参考基因组就构建好了,一般制作 psuedo-genomefa 就是为了消除变异带来的影响,部分其他参数可以到gatk官网查阅。另外 FastaAlternateReferenceMaker 使用简单的 indel ,当VCF文件包含复杂的位点时(complex substitutions),会忽略。
PS:
perEditor 用于分析phased SNPs/indels,意味着VCF文件中的 GT 是 0|1/1|1/1|2 ,而不是我们常见的 0/1 这种,明确知道变异是在母本还是父本染色体上,第一个是母本,第二个是父本,使用方式如下:
这里对参数进行一下解释:
allele 两个选项 mother|father ,代表等位基因来自于哪一个亲本;
indivicual ,整数,这个是因为有一些VCF文件中不止包含一个样本的突变信息,参数代表选择第几个样本的突变来进行创建 personalized genome , 1 代表第一个样本;
perEditor_ra ,允许用户把染色体重排信息考虑到创建个性化参考基因组中,一样只服务于phased的数据,另外还需要把涉及染色体重排的染色体放在一个目录下:
这里的 individual 和 allele 参数和 perEditor 是一样的含义;
rearrangements_tvcf ,染色体重排注释结果,vcf format version 41;
chr_length_tbed ,文件中包含基因组每一条染色体的长度,即核苷酸总数,格式如下:
centromeres_tbed ,文件中包含每条染色体的着丝粒位置的文件,着丝粒坐标用于确定新染色体的身份,当两条染色体由于重排而合并时,新染色体将按照着丝粒来源的名称来命名,继承两个着丝粒,其名称则包含两个亲本染色体的名称,文件格式如下:
bed文件和vcf文件位于同一个工作目录下,最终生成的新的染色体命名中添加了 new :
g2gtools 通过将SNP和indels整合到参考基因组中来创建自定义基因组,提取感兴趣的区域(例如外显子或转录本),并在两个基因组之间转换文件(bam,gtf,bed)的坐标。 与其他 liftover 工具不同, g2gtools 不会丢弃掉落在indel区域上的alignments。 版本02可以创建个人二倍体基因组。 新版本仍然将个人基因组坐标上的二倍体比对转换回参考基因组,因此我们可以比较种群样本之间的比对。
先激活virtual environment:
创建自定义的基因组,需要下列信息:
下面是 *** 作流程:
vcf2diploid 通过将phased的变异整合到参考基因组中,从vcf文件创建二倍体基因组。
samtools 、 bcftools 和 vcfutilspl 三个程序联合使用,从 BAM 文件开始 *** 作,到获得新的参考基因组:
FastaAlternateReferenceMaker
perEditor A tool to create personalized genome sequences
g2gtools creates custom genomes by incorporating SNPs and indels into reference genome
Welcome to g2gtools’ documentation
Personal Genome Constructor (vcf2diploid tool)
construct DNA sequence based on variation and human reference1、是deb格式,不是bed。。。
2、在Cydia中有一个目录,启动时会自动安装里面的deb文件。你在IPAD上装个iTools或者iFunbox,随便哪个都行,然后把从网上下载的deb文件用上述工具复制进去即可。具体目录是/private/var/root/Media/Cydia/AutoInstall/ 然后重启IPAD。deb文件会自动安装,安装后会自动删除。有时候重新启动一次还不行,多试几次即可。
3、最简单的方法!IPAD上装个iFile,然后把deb文件拷到IPAD里,随便哪个位置。拷完你用iFile找到他,点击后就可以直接安装程序,然后重启。
手打。。。求给分测序数据fa格式:一般为fagz文件
基因组参考序列fa格式:
第一行以@开头,之后为序列的标识符以及描述信息(与FASTA格式的描述行类似)
第二行为序列信息
第三行以+开头,之后可以再次加上序列的标识及描述信息(可选)
第四行为质量得分信息,与第二行的序列相对应,长度必须与第二行相同
GTF(Gene Transfer Format)格式是借鉴于GFF2格式,也被称为GFF25,大部分字段的定义是和GFF2相同的,只是每行的第九列必须带有如下四个域,具体为gene_id value; transcript_id value; 这样的设计是为了适应一个基因的多个转录本这种情况。GTF格式主要用来注释基因:
[上传失败(image-8d42c2-1536734614697)]
gff主要用来注释基因组
SAM(sequence alignment/map format):由标头注释部分和比对部分组成
bed(Browser Extensible Data):是ucsc 的genome browser的一个格式,描述注释的数据。bed有3个要求的字段(基本列)和9个额外的字段(附加列)
vcf(Variant Call Format):格式是用于表示突变信息的文本格式,可以用来表示SNV(single nucleotide variants),INDEL( insertions/deletions), CNV(copy number variants )and SV(structural variants)等。VCF格式同样是分为两大部分,一部分是注释描述信息,一部分是具体的突变信息,其中注释信息是以##开头的。
应用:
>
1,一般格式:PED/MAP
2,转置格式:TPED/TFAM
3,二进制格式:BED/BIM/FAM
几种格式之间可以相互转换。推荐使用BED/BIM/FAM这种格式,读取速度快。
bed 文件包含SNP数据,是二进制格式,不能由Notepad++等文本编辑器打开。
bim 文件包括SNP位置信息。
fam 文件包括家系表型信息,这两种文件都是文本格式。
其他格式转换命令: --recodeAD ,SNP编码成加性显性模式,以0、1、2编码SNP,NA为缺失值; --recode12 ,SNP编码为数字1或2,缺失值为0
参考推文:
>
欢迎分享,转载请注明来源:内存溢出

 微信扫一扫
微信扫一扫
 支付宝扫一扫
支付宝扫一扫
评论列表(0条)