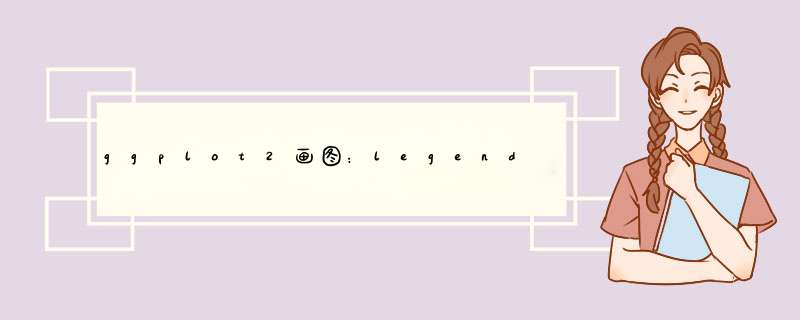
修改位置有两种方式,一种是直接给出四种位置中的一个:“left”,“top”, “right”, “bottom”, “none”。
另一种是给出数值型位置坐标:
修改多组的legend,导入mtcars数据集并画图:
欢迎关注~
#install.packages("reshape2")
#install.packages("ggpubr")
#引用包
library(reshape2)
library(ggpubr)
riskFile="risk.all.txt" #风险文件
estimateFile="TMEscores.txt" #肿瘤微环境打分文件
setwd("D:/※zixue/dead/ 140prgTME/47.estimateVioplot ")
#读取风险文件
Risk=read.table(riskFile, header=T, sep="\t", check.names=F, row.names=1)
#变为factor
Risk$risk=factor(Risk$risk, levels=c("low","high"))
#读取肿瘤微环境打分文件
score=read.table(estimateFile, header=T, sep="\t", check.names=F, row.names=1)
#提取 "StromalScore" "ImmuneScore" "ESTIMATEScore"
score=score[,1:3]
rownames(score)=gsub("(.*?)\\_(.*?)", "\\2", rownames(score))
score=score[row.names(Risk),,drop=F]
#数据合并
rt=cbind(Risk[,"risk",drop=F], score)
#将合并后的数据转换为ggplot2的输入文件
#melt
data=melt(rt, id.vars=c("risk"))
colnames(data)=c("Risk", "scoreType", "Score")
#绘制小提琴图
#x轴 肿瘤微环境打分类型;y轴 肿瘤微环境的打分
p=ggviolin(data, x="scoreType", y="Score", fill = "Risk",
xlab="",
ylab="TME score",
legend.title="Risk",
add = "boxplot", add.params = list(color="white"),#小提琴图中间,加箱线图
palette = c("blue","red"), width=1)#低风险蓝色,高风险红色
p=p+rotate_x_text(45)#x轴倾斜45度
#差异分析
p1=p+stat_compare_means(aes(group=Risk),
method="wilcox.test",
#<0.001 ***
symnum.args=list(cutpoints = c(0, 0.001, 0.01, 0.05, 1), symbols = c("***", "**", "*", " ")),
label = "p.signif")
#输出图形
pdf(file="vioplot.pdf", width=6, height=5)
print(p1)
dev.off()
欢迎分享,转载请注明来源:内存溢出

 微信扫一扫
微信扫一扫
 支付宝扫一扫
支付宝扫一扫
评论列表(0条)