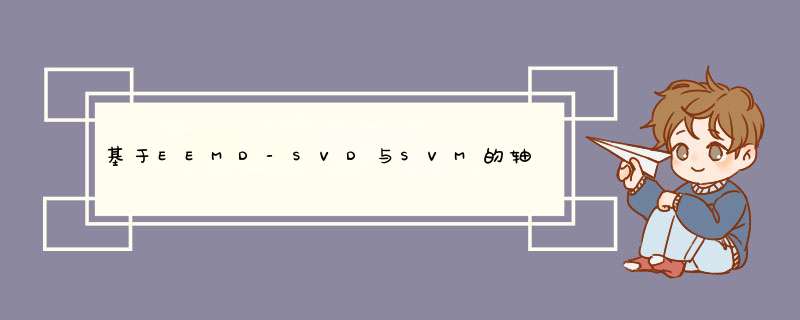
有一个矩阵A为m×n,,其中U 和 V的列分别叫做A的左奇异向量和 右奇异向量,Σ的对角线上的值叫做 A 的奇异值。
- 求 的特征值和特征向量,用单位化的特征向量构成 U
- 求 的特征值和特征向量,用单位化的特征向量构成 V
- 将 或者 的特征值求平方根,然后构成
数据为西储大学的轴承故障数据,所选数据为采样频率为12kHz、负载为0.75 kW(转速为1772 r/min) 的驱动端轴承数据。
包括正常数据,内圈故障直径 0.18,外圈( 6 点钟) 故障直径 0.36,滚动体故障直径0.53的四种数据。
import numpy as np
import heapq
from PyEMD import EEMD
import matplotlib.pyplot as plt
import matplotlib as mpl
import os
import glob
from sklearn.multiclass import OneVsRestClassifier
from sklearn.svm import SVC
import pandas
from sklearn.model_selection import train_test_split
from sklearn.metrics import classification_report, confusion_matrix, recall_score
from sklearn.model_selection import GridSearchCV
from sklearn.model_selection import cross_val_score
2.4、进行eemd分解,并将分解的IMF分量通过计算峭度值进行筛选,选取大小前面六个IMF分量,构成奇异矩阵,再进行奇异分解,最后将奇异值构成特征矩阵作为输入训练集。
for i in range(300,400):
y = data1[i]
eemd = EEMD()
eIMFs = eemd.eemd(y)
nIMFs = eIMFs.shape[0]
# print(nIMFs)
kurts = []
for j in range(nIMFs):
data = eIMFs[j]
N = len(data)
df_kurt = N*np.sum((data-np.mean(data))**4)/(np.sum((data-np.mean(data))**2))**2-3
kurts.append(df_kurt)
# print(kurts)
tmp = zip(range(len(kurts)), kurts)
large5 = heapq.nlargest(6, tmp, key=lambda x:x[1])
# print(large5)
indexs = []
for k in range(6):
index = large5[k][0]
indexs.append(index)
# print(indexs)
# print(eIMFs[indexs[0]])
juzhen1 = np.array([[eIMFs[indexs[0]]],[eIMFs[indexs[1]]],[eIMFs[indexs[2]]],[eIMFs[indexs[3]]],[eIMFs[indexs[4]]],[eIMFs[indexs[5]]]])
juzhen = np.mat(juzhen1)
U,s,V = np.linalg.svd(juzhen)2.5、用支持向量机进行分类
model1 = SVC(C=2, kernel='linear', decision_function_shape='ovr',gamma=1)
clf1 = model1.fit(x_train, y_train)
model2 = SVC(C=2, kernel='rbf', gamma=1)
clf2 = model2.fit(x_train, y_train)
# 使用poly多项式核函数
model3 = SVC(kernel='poly',C=2,gamma=1)
clf3 = model3.fit(x_train, y_train)
# 使用sigmoid神经元激活核函数
model4 = SVC(kernel='sigmoid')
clf4 = model4.fit(x_train, y_train)
# 打印使用不同核函数进行分类时,训练集和测试集分类的准确率
# print("rvc-训练集:", model.score(data_train_x, data_train_y))
# print("rvc-测试集:", model.score(data_test_x, data_test_y))
print("linear线性核函数-训练集:", model1.score(x_train, y_train))
print("linear线性核函数-测试集:", model1.score(x_test, y_test))
print("rbf径向基核函数-训练集:", model2.score(x_train, y_train))
print("rbf径向基核函数-测试集:", model2.score(x_test, y_test))
print("poly多项式核函数-训练集:", model3.score(x_train, y_train))
print("poly多项式核函数-测试集:", model3.score(x_test, y_test))
print("sigmoid神经元激活核函数-训练集:", model4.score(x_train, y_train))
print("sigmoid神经元激活核函数-测试集:", model4.score(x_test, y_test))
# 绘制混淆矩阵
print('预测标签', predictions)
print('真实标签', y_test)
classes = list(map(int, y_test))
# 排序,准确对上分类结果
classes.sort()
# 对比,得到混淆矩阵
confusion = confusion_matrix(y_test, predictions)
# 热度图,后面是指定的颜色块,gray也可以,gray_x反色也可以
plt.imshow(confusion, cmap=plt.cm.Blues)
# 这个东西就要注意了
# ticks 这个是坐标轴上的坐标点
# label 这个是坐标轴的注释说明
indices = range(len(confusion))
# 坐标位置放入
# 第一个是迭代对象,表示坐标的顺序
# 第二个是坐标显示的数值的数组,第一个表示的其实就是坐标显示数字数组的index,但是记住必须是迭代对象
plt.xticks(indices, ['ball', 'inner', 'normal', 'outer'])
plt.yticks(indices, ['ball', 'inner', 'normal', 'outer'])
# 热度显示仪?就是旁边的那个验孕棒啦
plt.colorbar()
# 就是坐标轴含义说明了
plt.xlabel('y_test')
plt.ylabel('predictions')
plt.title('混淆矩阵')
# 显示数据,直观些
for first_index in range(len(confusion)):
for second_index in range(len(confusion[first_index])):
plt.text(first_index, second_index, confusion[first_index][second_index])
# 显示
plt.show()
3、完整代码
mpl.rcParams['font.sans-serif'] = ['KaiTi'] # 保证正常显示中文
mpl.rcParams['font.serif'] = ['KaiTi'] # 保证正常显示中文
mpl.rcParams['axes.unicode_minus'] = False # 保证负号正常显示
path = r'E:/eemd-svd/shuju/'
def read_txt(path):
cate = [path + x for x in os.listdir(path) if os.path.isdir(path + x)]
imgs = []
labels = []
for idx, folder in enumerate(cate):
for im in glob.glob(folder + '/*.txt'):
img = np.loadtxt(im)
imgs.append(img)
labels.append(idx)
return np.asarray(imgs, np.float32), np.asarray(labels, np.int32)
data1, label = read_txt(path)
print(label)
S0 = []
S1 = []
S2 = []
S3 = []
S4 = []
S5 = []
for i in range(300,400):
y = data1[i]
eemd = EEMD()
eIMFs = eemd.eemd(y)
nIMFs = eIMFs.shape[0]
# print(nIMFs)
kurts = []
for j in range(nIMFs):
data = eIMFs[j]
N = len(data)
df_kurt = N*np.sum((data-np.mean(data))**4)/(np.sum((data-np.mean(data))**2))**2-3
kurts.append(df_kurt)
# print(kurts)
tmp = zip(range(len(kurts)), kurts)
large5 = heapq.nlargest(6, tmp, key=lambda x:x[1])
# print(large5)
indexs = []
for k in range(6):
index = large5[k][0]
indexs.append(index)
# print(indexs)
# print(eIMFs[indexs[0]])
juzhen1 = np.array([[eIMFs[indexs[0]]],[eIMFs[indexs[1]]],[eIMFs[indexs[2]]],[eIMFs[indexs[3]]],[eIMFs[indexs[4]]],[eIMFs[indexs[5]]]])
juzhen = np.mat(juzhen1)
U,s,V = np.linalg.svd(juzhen)
print(s)
S0.append(s[0])
S1.append(s[1])
S2.append(s[2])
S3.append(s[3])
S4.append(s[4])
S5.append(s[5])
np.savetxt('E:/eemd-svd/outer_svd0.txt',S0)
np.savetxt('E:/eemd-svd/outer_svd1.txt',S1)
np.savetxt('E:/eemd-svd/outer_svd2.txt',S2)
np.savetxt('E:/eemd-svd/outer_svd3.txt',S3)
np.savetxt('E:/eemd-svd/outer_svd4.txt',S4)
np.savetxt('E:/eemd-svd/outer_svd5.txt',S5)
x = []
y = []
data = open('E:/eemd-svd/eemd-svd.csv',encoding='UTF-8-sig')
for line in data.readlines():
lineArr = line.strip().split(',')
# data_train.append(lineArr)
x.append(lineArr[1:7])
y.append(lineArr[0])
x_train,x_test,y_train,y_test = train_test_split(x,y,test_size=0.3)
print(y_test)
model1 = SVC(C=2, kernel='linear', decision_function_shape='ovr',gamma=1)
clf1 = model1.fit(x_train, y_train)
model2 = SVC(C=2, kernel='rbf', gamma=1)
clf2 = model2.fit(x_train, y_train)
# 使用poly多项式核函数
model3 = SVC(kernel='poly',C=2,gamma=1)
clf3 = model3.fit(x_train, y_train)
# 使用sigmoid神经元激活核函数
model4 = SVC(kernel='sigmoid')
clf4 = model4.fit(x_train, y_train)
# 打印使用不同核函数进行分类时,训练集和测试集分类的准确率
# print("rvc-训练集:", model.score(data_train_x, data_train_y))
# print("rvc-测试集:", model.score(data_test_x, data_test_y))
print("linear线性核函数-训练集:", model1.score(x_train, y_train))
print("linear线性核函数-测试集:", model1.score(x_test, y_test))
print("rbf径向基核函数-训练集:", model2.score(x_train, y_train))
print("rbf径向基核函数-测试集:", model2.score(x_test, y_test))
print("poly多项式核函数-训练集:", model3.score(x_train, y_train))
print("poly多项式核函数-测试集:", model3.score(x_test, y_test))
print("sigmoid神经元激活核函数-训练集:", model4.score(x_train, y_train))
print("sigmoid神经元激活核函数-测试集:", model4.score(x_test, y_test))
# 使用decision_function()可以查看决策函数
print(model2.decision_function(x_train))
# 使用predict()可以查看预测结果
print(model2.predict(x_train))
# 输出混淆矩阵
predictions = model2.predict(x_test)
cm = confusion_matrix(y_test, predictions)
print(cm)
# 5折交叉验证
score_SVM = cross_val_score(model2, x_test, y_test, cv=5) # 五折交叉验证
print(score_SVM)
print('score均值', np.mean(score_SVM)) # 五折交叉验证的均值
# 绘制混淆矩阵
print('预测标签', predictions)
print('真实标签', y_test)
classes = list(map(int, y_test))
# 排序,准确对上分类结果
classes.sort()
# 对比,得到混淆矩阵
confusion = confusion_matrix(y_test, predictions)
# 热度图,后面是指定的颜色块,gray也可以,gray_x反色也可以
plt.imshow(confusion, cmap=plt.cm.Blues)
# 这个东西就要注意了
# ticks 这个是坐标轴上的坐标点
# label 这个是坐标轴的注释说明
indices = range(len(confusion))
# 坐标位置放入
# 第一个是迭代对象,表示坐标的顺序
# 第二个是坐标显示的数值的数组,第一个表示的其实就是坐标显示数字数组的index,但是记住必须是迭代对象
plt.xticks(indices, ['ball', 'inner', 'normal', 'outer'])
plt.yticks(indices, ['ball', 'inner', 'normal', 'outer'])
# 热度显示仪?就是旁边的那个验孕棒啦
plt.colorbar()
# 就是坐标轴含义说明了
plt.xlabel('y_test')
plt.ylabel('predictions')
plt.title('混淆矩阵')
# 显示数据,直观些
for first_index in range(len(confusion)):
for second_index in range(len(confusion[first_index])):
plt.text(first_index, second_index, confusion[first_index][second_index])
# 显示
plt.show()注:其中分类的数据eemd-svd.csv需要自己生成
欢迎分享,转载请注明来源:内存溢出

 微信扫一扫
微信扫一扫
 支付宝扫一扫
支付宝扫一扫
评论列表(0条)