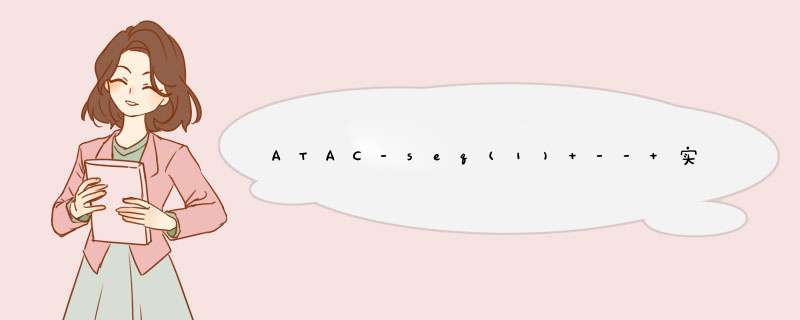
染色质关闭:压缩DNA
这个过程像我们将文件压缩为zip或者rar的压缩包,减少它的占用空间。
染色质开放:解压DNA
这个过程类似于我们要查看刚刚压缩包里的文件,我们需要解压后才能查看到文件里的内容。
ATAC-seq,全称Assay for Transposase-Accessible Chromatin with high throughput sequencing,是2013年由斯坦福大学William J Greenleaf和Howard Y Chang实验室开发的用来研究染色质可及性(通常也理解为染色质的开放性)的方法
1 原理
ATAC-seq 是通过使用高通量测序技术对转座酶Tn5可接近性核染色质区域进行分析的一种新型表观遗传学研究技术。该技术通过转座酶对某种特定状态下开放的核染色质区域进行切割,获得在该状态下基因组中所有活跃转录的调控序列,再通过聚类分析,挖掘这些开放位点的潜在结合转录因子,结合基因表达水平数据,发现关键的调控转录因子。
这个技术用到了一个转座酶Tn5:DNA转座是一种由DNA转座酶介导,把DNA序列从染色体的一个区域插入到另外一个区域的现象,类似于“剪切粘贴”。这个过程,也是需要插入位点的染色质是开放的,否则就会被一大坨高级结构给卡住。
既然转座酶Tn5容易结合在开放染色质上,只要人为地将将NGS接头连接到转座酶,携带这些接头的转座酶(如下图带着红色蓝色测序标签的转座酶Tn5)进入细胞核后,在染色质开放区域各种切切切,使染色质断裂并将这些接头插入到开放的染色质区域中,这样我们裂解细胞、破碎DNA后,利用已知序列的测序标签进行NGS测序,就知道哪些区域是开放染色质了。
2 应用
染色质开放性图谱绘制
找调控生物学过程的关键转录因子
找哪个转录因子调控了研究的基因
找转录因子调控的靶基因
3 技术限制
Tn5通过插入剪断DNA 并将测序接头连接到剪断的两个DNA 片段的末端,因此对于一个DNA 片段而言,其两端的接头连接是随机的,这便导致同一片段两端的接头有50%的概率是同一接头。而只有连接不同接头的片段才可用于富集扩增及测序,因此,有一半的片段无法利用;
大量剪断的DNA 由于片段过大,无法进行PCR富集;
Tn5 的活性受反应溶液的组成及反应条件影响,仍然需要优化以便提高剪切效果;
ATAC-seq在植物细胞中存在以下难点:细胞壁的存在,叶绿体、线粒体等细胞器的污染,缺少稳定遗传的细胞系;
分享给你们。
ATAC-seq:染色质开放性测序技术 - 知乎 (zhihucom)
盗它的几张图吧,自己你不懂的时候也可以随时翻出来看看。嘿嘿。
真核生物的核DNA并不是裸露的,而是有蛋白质即组蛋白与之相结合的。DNA一圈一圈地缠绕在组蛋白上,形成串珠式的结构。而这样的结构还能够进一步折叠、浓聚,并在其他架构蛋白的辅助下,形成染色体。
打开的染色质,就叫开放染色质(open chromatin)。而染色质一旦打开,就允许一些调控蛋白(比如转录因子和辅因子)跑过来与之相结合。而染色质的这种特性,就叫做染色质的可进入性(chromatin accessibility)。
DNA转座,是一种把DNA序列从染色体的一个区域搬运到另外一个区域的现象,由DNA转座酶来实现。这种转座插入DNA,也是需要插入位点的染色质是开放的,否则就会被一大坨高级结构给卡住。
那么,如上图a,我们只要人为地,将携带已知DNA序列标签的转座复合物(即带着红色蓝色测序标签的转座酶Tn5),加入到细胞核中,再利用已知序列的标签进行PCR后测序,就知道哪些区域是开放染色质了。而这也就是ATAC-seq的原理。
ATAC-seq出来的结果,和传统方法出来的结果具有很强的一致性,同时也和基于组蛋白修饰marker的ChIP-seq有较高的吻合程度。也就是说,ATAC-seq中的peak,往往是启动子、增强子序列,以及一些反式调控因子结合的位点。
相比起来,ATAC-seq的重复性,比MNase-seq和DNase-seq的更强, *** 作起来也更加简单,而且只需要很少的细胞/组织量,同时出来的信号更加漂亮。目前已经是研究染色质开放性首选的技术方法。
在准备NGS文库的时候,会有用到转座酶Tn5, Nextera DNA Library Preparation Kit ,比如ATAC-seq就有用到这个Tn5。转座酶携带有特定的序列称为 转座子Transposon 。
下面是ATAC-seq的工作原理,想必听说过ATAC-seq的,对这个图都会再熟悉不过了。
Tn5是kit里面带有的,没什么大不同。ATAC-seq 的barcoded primers (adaptors)会有一些不同,接下来,看下图中的每段序列对应的sequnce都是什么。
下面图例,Nextera tn5用来给基因组DNA加adaptors。
下面的就是准备ATAC-seq时候需要用到的index primer (adaptor),和Nextera DNA library prep kit里的有一点类似,但是也很不同。Ad1_noMX是Forward 引物,没有index (barcode),所以只有一个。Reversed引物有24个Ad2(1-24),所以有24组不同的index (barcode)序列。这个index序列是和Nextera一样的。我涂红色部分就是index序列的反向反义序列,每个index序列是不同的8个碱基,这个和Nextera的i7(部分一样,请自己对照)是一样的。
i5 sequence: ATAC-seq的Adaptor1 里没有barcode
i7 sequence:
有两种Adaptors,分别为Adaptor1 (50bp) 和Adaptor2 (53bp)。
其中Adaptor1(5’ illumina Primer1 sequence (29bp)+ Tn5 Read1 sequence(14bp)(再延伸到ME里面7bp)3’),Adaptor2(5’ illumina Primer2 sequence(24bp)+ barcode sequence (8bp)+ Tn5 Read2 sequence (15bp)(再延伸到ME里面6bp)3’)
这样算来,ATAC-seq library的长度=左面(Adaptor1 长度 +剩余部分Tn5的ME(12 bp_TATAAGAGACAG))+ open chromatin 的DNA长度+右面(剩余部分Tn5的ME(13bp_GTATAAGAGACAG)+ Adaptor2 长度)=open chromatin 的DNA长度 + 128bp。
所以,在ATAC-seq的libraries中 mono nucleosome的ATAC-seq library长度大概是(单核小体146bp+核小体free region)+两端的adaptor及Tn5最里面部分ME序列长度(50bp+12bp+13bp+53bp)=274bp+free region,也就是bioanalyzer里面看到的第二个peak。图中显示大概是340bp左右。所以free region应该是330bp-274bp=56bp 。
那么会问,对不对呢?看下bioanalyzer的第一个peak,显示是182bp,这个peak代表的是nucleosome free region的ATAC-seq library,所以是nucleosome free region序列长度 + 两端的adaptor及Tn5部分ME序列长度(50bp+12bp+13bp+53bp)=182, 所以,不难得出同样的结果,nucleosome free region序列长度是54bp。差不多哦
以此类推,第三个peak代表de-nucleosome , 500bp到550 bp之间,是不是等于两个核小体长度加上核小体空隙序列再加上两个adaptor, 2 146bp + 2 55bp + 128bp= 530bp (为什么55bp也要乘2?答:多出来一个核小体当然就多出来一份核小体空隙)
nucleosome free region, mono-nucleosome, de-nucleosome and try-nucleosome
“GAP不是服装品牌,而是个坑”。
所以需要在PCR第一步,需要5min的72摄氏度来填坑。
还要注意,就是在tagmentation的时候会出现三种产物,上面的图只是其中一种。
哪里说的不对欢迎纠正,请留言。
参考:
>
欢迎分享,转载请注明来源:内存溢出

 微信扫一扫
微信扫一扫
 支付宝扫一扫
支付宝扫一扫
评论列表(0条)