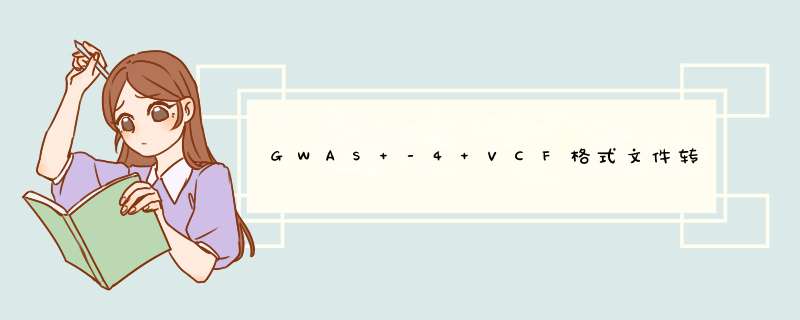
plink1.9版本支持转化为VCFv4.2格式
plink2.0版本支持转化为VCFv4.3格式
两个版本用到的命令不一样
对于plink1.9版本,转化为vcf文件的命令行为:
生成的vcf为4.2版本
对于plink2.0版本,转化为vcf文件的命令行为:
生成的vcf为4.3版衫宽本
参考: https://www.cnblogs.com/chenwenyan/p/8574237.html
链接: https://www.jianshu.com/p/8b4e7b3b7f5e
vcf 转为 ped/map
vcftools --vcf snp.vcf --plink --out snp
plink --vcf snp.vcf --recode --out snp
ped和map文件是Plink的基本格式。
ped文件包含以下几列:
第一列:Family ID。
第二列:Individual ID。自然群体这列和Family ID是一样的。
第三列:Paternal ID。未提供信息的话这列为0。
第四列:Maternal ID。未提供信息的话这列为0。
第五列:Sex。未提供信息的话这列为0。
第六列:Phenotype。一般来说,直接拿vcf转换的话这列为-9,也就是缺失。
第七列开始就是个体在每个标记位点的基因型。
map文件包含以下几列:
第一列:染色体编号。
第二列:SNP编号。
第三列:遗传距离。未提供信息的话这列为0。
第四列:物理位置。
ped/map 与 tped/tfam 格式互换
plink --file snp --recode --transpose --out snp_test
plink --tfile snp_test --recode --out snp
ped/map 与 bed/bim/fam互换
plink --file snp --make-bed --out snp_test
plink --bfile snp_test --recode --out snp
tped/tfam 与 bed/bim/fam互换
plink --tfile snp --make-bed --out snp_test
plink --bfile snp_test --recode --transpose --out snp
bed/bim/fam 转为 vcf
plink --bfile snp --export vcf --out snp_test
常用的Plink格式转换就是这些,大家可以根据自己实际需要相互转换。
因为PLINK默认的设置是人的染色体简粗, 所以动物中,我们应该设置
--chr-set 19 # 猪
已有的选择:
--cow
--dog
--horse
--mouse
--rice
--sheep
参考:或咐亮 https://zhuanlan.zhihu.com/p/109071456
我现在的问题是这样的vcf文件转plink的格式
方法一
vcftools
出错这样
方法二plink
都是一样的结果
就无语
之前我的文件有这些
现在是这些
然后看到了这些
然后发现文件又是临时文件
说明又出错了
所以,现在或许又要解决那一行的无效染色体
然后我就使用了--allow-extra-chr
哈,又回到了族慎渗最初的起点
可是我看意思,plink2不应该有这个问题啊
应该是 --vcf-half-call的格孝友式不对
然后换回plink1
重看官网
以及 https://www.jianshu.com/p/38e603979334?utm_campaign=maleskine&utm_content=note&utm_medium=seo_notes&utm_source=recommendation
发现格式
发现原来是因为少了空格
终于成功,哭了
https://www.jianshu.com/p/dc82fcbe3cda 然后意识到自己搜索兆脊关键词有问题
这回终于对了
但问题是第一种转的,不知道应该是哪一种
plink官网: http://zzz.bwh.harvard.edu/plink/plink功能:数据管理、质控、人群分层检测、关联分析-SNP、多marker预测、haplotype分析、拷贝升做数变异分析、异位显性分析、关联分析-基因、基因环境互作分析、meta分析等。
第一列:家庭编号family id
第二列:个体编号individual id
第三列:父亲编号paternal id
第四列:母亲编号maternal id
第五列:性别(1 male 2 female)
第六列:基因型genotype
第一列:染色体编号1-22 X Y 0(unplaced)
第二列:SNP rs编号
第三列:morgen遗传距离
第四列:碱基对位置bp
PLINK可以在ped和map文件谨虚的基础上通过创建binary ped file (.bed)来节省运行时间和存储空间。这种格式的文件将pedigree/phenotype information存储在.fam文件中,并且通过创建一个.bim文件来储存map信息(包括allele name等)。
在plink中可以通过以下命令创建binary ped文件:
以上命令行将创建出以下三个文件:
plink.bed ( binary file, genotype information )
plink.fam ( first six columns of mydata.ped )
plink.bim ( extended MAP file: two extra cols = allele names)
自行创建表型文件pheno.txt,可在其中存入多种表型或协变量。
前两列必须为FID和IID,后面是表型:
第一列:家庭编号
第二列:个人编号
其他列:表型数据
案例:
格式与表型文件相同。
案例:
方法:
结果文件1:个体.imiss
第一列:家庭编号
第二列:个体编号
第三列:缺失SNP数
第四列:number of non-obligatory missing genotype
第五列:缺失SNP占比
结果文件2:SNP.lmiss
第一列:SNP编号
第二列:染色体编号
第三列:缺失该SNP的个体数
第四列:number of non-obligatory missing genotype
第五列:缺失该SNP的个体数占比
方法:
结果文件:file.hwe
方法:
结吵晌衡果文件:
说明:设置阈值如果超过阈值则删除对应的SNP或个体
结果文件:
结果文件:outlier.nearest
结果文件:plink.assoc
结果文件:plink.fisher
结果文件:plink.model
通过回归矫正协变量。
结果文件:plink.assoc.linear/logistic
更多阅读:
1 GWAS 学习笔记 | 从理论到实践
2 全基因组关联分析(GWAS) — 群体结构
欢迎分享,转载请注明来源:内存溢出

 微信扫一扫
微信扫一扫
 支付宝扫一扫
支付宝扫一扫
评论列表(0条)