LDA是监督学习的降维、分类技术,可以用一句话概括思想:投影后类内方差最小,类间方差最大。
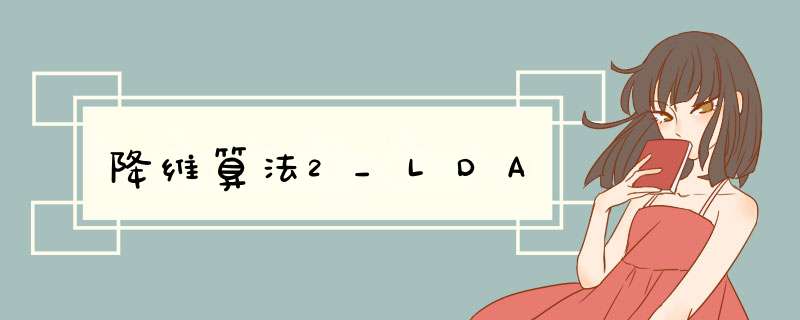
如下图所示,给定数据样例集,设法将样例投影到直线上,使得同类样例投影点尽可能近、异类样例的投影点尽可能远离:
下图的两种投影方式,那一种满足我们的标准?
右图,原因是投影后,蓝色和红色数据各自较为集中,且类别之间的距离明显。(左图在边界处数据混杂
LDA如何用来分类?
在对新的样本进行分类时,将其投影到同样的投影线上,再根据投影点的位置来确定样本的类别。
目标:求解投影向量 ,二维情况下 ,
,shape:(2,m)表示样本矩阵,即2个维度,m个样本。每一列表示一个样本,每一行表示该样本在这一维度上的取值。例如: 中的第一列表示第一个样本在第一个维度上的取值,在第二个维度上的取值为,以此类推。
对第个样本 :
图中的y可计算,对于样本在投影向量上的投影结果的值为: , 可以理解为这条过原点直线的单位方向向量, 为 的夹角。
给定两个类别:
X1 : shape:(m,2)一般将样本按行排列,每列表示维度,可以理解为 的转置,表示m个样本,2两个维度,图中每个点表示
X2 : 描述同X1
均值向量 : shape:(2,1) 将m个样本取每个维度的均值,按列排列,得到均值均值向量
协方差矩阵 : shape:(2,2) (维度x维度)
样本中心在直线上的投影 :shape:(1,2)x(2,1)= (1,1)常量
同类样本投影点的协方差: :shape:(1,2)x(2,2)x(2,1) = (1,1)常量 ,表示将同类样本投影后,两个维度的协方差。
对于n维情况,由于我们是多维向低维投影,则此时投影到的低维空间就不是一条直线,而是一个超平面了。假设我们投影到的低维空间的维度为d,对应的基向量为 ,基向量组成的矩阵为
此时同类样本投影点的协方差 shape: (d,n)x(n,n)x(n,d)=(d,d) ,表示投影后dxd的协方差矩阵。
程序
输入:数据集 ,,其中任意样本 为n维向量, 为样本对应的标签,降维到的维度d。
输出:降维后的样本集
1) 计算类内散度矩阵
2) 计算类间散度矩阵
3) 计算矩阵
4) 计算的 最大的 个特征值对应的个特征向量 ,得到投影矩阵
5) 对样本集中的每一个样本特征 ,转换为新的样本
6) 得到输出样本集
实际上LDA除了可以用于降维以外, 还可以用于分类 。一个常见的LDA分类基本思想是假设各个类别的样本数据符合高斯分布,这样利用LDA进行投影后,可以利用极大似然估计计算各个类别投影数据的均值和方差,进而得到该类别高斯分布的概率密度函数。 当一个新的样本到来后,我们可以将它投影,然后将投影后的样本特征分别带入各个类别的高斯分布概率密度函数,计算它属于这个类别的概率,最大的概率对应的类别即为预测类别 。
相同点:
1)两者均可以对数据进行降维。
2)两者在降维时均使用了矩阵特征分解的思想。
3)两者都假设数据符合高斯分布
不同点
1LDA是有监督的降维方法,而PCA是无监督的降维方法n
2LDA降维最多降到类别数k-1的维数,而PCA没有这个限制。
3LDA除了可以用于降维,还可以用于分类。
4LDA选择分类性能最好的投影方向,而PCA选择样本点投影具有最大方差的方向。这点可以从下图形象的看出,在某些数据分布下LDA比PCA降维较优。
当然,某些某些数据分布下PCA比LDA降维较优,如下图所示:
维基百科介绍:主成分分析(英语:Principal components analysis,PCA)是一种分析、简化数据集的技术。主成分分析经常用于减少数据集的维数,同时保持数据集中的对方差贡献最大的特征。
说了和没说一样……我们还是通过一个简单的案例引出PCA的作用吧。
如果我们在6个小鼠样本中检测一个基因 Gene1 的表达
我们很容易看出来,基因 Gene1 在小鼠1-3中表达比较相似,而在小鼠4-6中表达比较相似
如果同时检测两个基因
我们可以将不同小鼠样本标记在二维坐标轴中,并且看出小鼠1-3的整体表达比较相似,而小鼠4-6的整体表达比较相似
将基因数目扩增到3个时候,我们依然可以通过三维坐标轴标记出不同样本的分布
但是如果将基因数目增加到4个或4个以上时候,很难继续增加坐标轴的维度来绘图(思维空间已经超出一般人的认知了)。
所以 我们可以通过PCA的降维方法来处理这种4维或者多维数据,将其绘制为二维图像来比较不同样本之间的关系 。
PCA是如果进行降维的呢?
首先我们只检测6个不同小鼠的2个基因,那么我们可以分别计算出所有小鼠 Gene1 和 Gene2 的平均值(红色叉号)。根据这些均值,可以获得所有数据的中心(蓝色叉号)。
然后我们将数据整体移动,数据的中心于原点重合。虽然所有的数据点都移动了,但是每个数据点的相对距离没有改变,只是数据的中心变为原点 (0,0) 。
接下来,我们会绘制一条通过原点的直线。这条直线可以360°旋转,直至同数据匹配最佳。
那么如何判断 最佳匹配 呢?这个标准是——
PCA将所有的数据点投射到这条直线上,并且计算这些数据点投射到直线上的距离(使这些距离最小)和投射点到原点的距离(使这些距离最大)
实际上计算c会简单一些,所以PCA一般是通过计算所有数据点到原点距离平方和sum( )最大值来寻找最优解。
如下图所示,分别将这6个样本的投射点到原点距离标记为d1,d2,,d6,然后计算这些点的平方和,这些平方和也被称为SS距离,即 。
获得SS最大值时的这条线,被称为 PC1 。假设PC1这条线的斜率为025,这意味着每当我们在 Gene1 前进4个单位时,PC1上的数据点在 Gene2 上就增加1个单位。
这也意味着这几个样本在 Gene1 上更加分散,而在 Gene2 上分散程度较小。
根据鸡尾酒配方来思考PC1,为生成PC1,我们加入了4份 Gene1 和1份 Gene2 ,这也说明在描述数据的分散程度方面, Gene1 更加重要。
在数学上,这种鸡尾酒配方被称为 Gene1 和 Gene2 的线性组合,或也可以说“PC1是几个变量的线性组合”。
在PCA中,将SS称为PC1的 特征值 (Eigenvalue);PC1特征值的平方被称为PC1的 奇异值 (Singular Value)。
根据上面的假设,PC1的斜率为025,如果下图的红色箭头是一个长度单位,那么它是有097个Gene1和0242个Gene2构成,所以 被称为 特征向量 (Eigenvector)或奇异向量(Singular vector)。
而每个基因的比例,被称为PC1的 载荷分数 (loading score)
在二维坐标中,PC2是一条通过原点,并且与PC1 垂直 的直线。
很容易计算出PC2的斜率为-4,那么PC2就是由-0242个Gene1和097个Gene2构成了。
同时,我们也可以计算出PC2的特征值。
旋转坐标轴,将PC1水平,PC2垂直。黑色叉表示原始的样本6,那么在新的坐标系中,Sample6的分布如下图所示。
各个主成分的变异度(Variation)计算,方法是SS除以样本减1。
假如上面那个案例中PC1变异度为15,PC2的变异度为3,那么总变异度为18
因此PC1在总变异中所占的比值为83%,PC2占的总变异为17%。
如果我们有3个基因,根据前面描述的步骤,分别找出PC1、PC2(垂直于PC1)和PC3(同时垂直于PC1和PC2),
同时也可以计算出各个主成分的变异度。
在上面的案例中,只使用PC1和PC2可以解释94%的变异度,所以我们只保留PC1和PC2最终绘图
这样,我们就获得了最终降维之后的结果。
在进行PCA降维之前,需要确保所有数据处于同一标准下。
例如下面这组数据,Math分数是按照百分制统计,而Reading分数是按照十分制统计。
那么我们在计算PC1时可能会得到PC1由099个Math和01个Reading组成,但是这仅仅是由于Math分数本身就是Reading分数的10倍。
所以我们需要 首先将每一个变量除以其所在组的标准差进行标准化处理 。
上面的那个案例中,我们只有两组观测数据。
绘制完PC1和PC2后,还可以绘制出PC3吗?
根据之前的讲述,PC3是同时垂直于PC1和PC2的。
那么绘制PC3时,基于已有的数据,先去寻找垂直于PC1的直线,只会得到PC2。
寻找垂直于PC2的直线,也只会得到PC1。
所以无法绘制出PC3。
如果上面的案例中,Math和Reading是100%相关的
绘制完PC1后,绘制一条垂直于PC1的PC2时发现,所有的数据投射在PC2的点都是原点,即特征值为0。
这时,PC1能够100%解释所有的变异。所以此时仅有PC1一个主成分。
如果学生数目减少为2,那么数据的分布在二维平面只有2个点,
2个点仅可以构成一条直线,所以PC2的特征值必然为0。
即使增加一个观测指标Gym,如果只有两个学生的话,最终数据在三维空间只有两个点,所以也只会有一个主成分PC1。
扩展一下,如果数据变为3个学生和3个观测指标,最终会有几个主成分呢?
3个学生会产生3个数据点,3点构成一个平面(平面是二维的),所以PC3的特征值必然为0。
因此会产生2个主成分PC1和PC2。
所以, 一组数据进行降维分析时,主成分数最终等于变量数目或样本数目(二者较小的那个),但是上限是特征值大于0的PC数 。
如果每次只取一个波段进行计算的话,直接
for m = 1:200DR=[salinas_corrected(:,:,m)];
end
就可以了
每次使用DR进行计算,而DR的维数就是500×300。
以上就是关于降维算法2_LDA全部的内容,包括:降维算法2_LDA、PCA降维原理、matlab 三维矩阵降维等相关内容解答,如果想了解更多相关内容,可以关注我们,你们的支持是我们更新的动力!
欢迎分享,转载请注明来源:内存溢出

 微信扫一扫
微信扫一扫
 支付宝扫一扫
支付宝扫一扫
评论列表(0条)