目前,用于Hi-C辅助基因组组装的软件有LACHESIS、SALSA2、3D-DNA、ALLHiC等,包括这2年发的hic_hiker等, 这些软件在基因组组装方面各有优劣。SALSA2和3D-DNA虽不需预先提供染色体数目即可进行互作分析,但在可 *** 作性和实用性上都有一定的局限性。LACHESIS作为分析Hi-C数据的经典工具,文章认可度较高,但其在多倍体基因组组装方面表现欠佳。前面也试过AllHiC来挂载多倍体,但是有些简单的物种挂载过程中好像有点不如意的区域,所以再尝试一下去其它工具的结果。
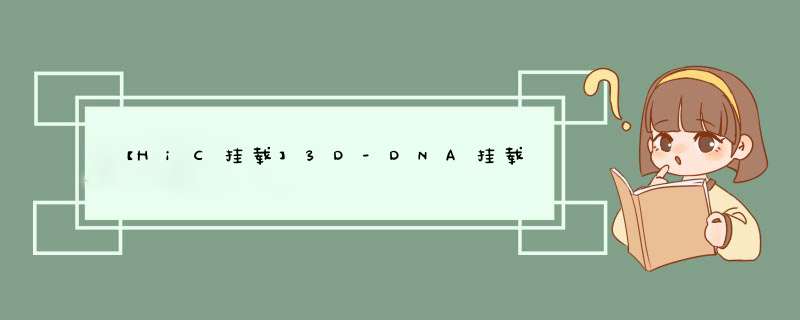
使用3D-DNA做基因组组装的整体流程如下图,分别为组装,Juicer分析Hi-C数据,3D-DNA进行scaffolding,使用JBAT对组装结果进行手工纠正,最终得到准染色体水平的基因组。
=====安装=====
在安装之前,确保服务器上有了下面这些依赖软件工具
LastZ(仅在杂合基因组的二倍体模式下使用)
Java >= 17
GNU Awk >= 402
GNU coreutils sort > 811
Python >= 27
scipy, numpy, matplotlib
GNU Parallel >=20150322 (不必要,但是强力推荐)
bwa
我们需要安装两个软件,一个是3D-DNA,另一个是juicer。
CPU版本的juicer:
git clone >dna文件解析错误怎么办,原因如下:
第一种原因,这些信息在服务器底层数据库存储时出现了格式错误,此时需要管理员执行数据库周期性的archive *** 作,纠正这些格式错误。
第二种原因,客户端使用的服务器检索程序编写有故障,或者不能处理所有的特殊格式。这种情况需要增强检索查询。
2在运行窗口中,输入cmd,按下回车键,打开命令窗口;
3在命令窗口输入命令ipconfig /all ,按下回车键,刷出一堆字,找打NDS服务器后面的ip地址就是了。
4将得到的DNS地址写进电脑;
5点击任务栏上的网络,打开网络和共享中心;
6在导航栏找到更改适配器设置选项,打开它;
7右键单击你要设置dns的网络,然后再打开的右键菜单中,选择属性;
8在属性面板中,选中IP4,然后点击下面的属性;
9在属性面板中,切换到常规选项下,勾选使用下面的DNS服务器地址,输入DNS地址,然后点击确定按钮,设置完成。
欢迎分享,转载请注明来源:内存溢出

 微信扫一扫
微信扫一扫
 支付宝扫一扫
支付宝扫一扫
评论列表(0条)